La thématique de recherche de l’équipe consiste à caractériser les mécanismes moléculaires impliqués dans les interactions entre cellules hématopoïétiques et stromales au sein des niches médullaires.
L’hématopoïèse est le mécanisme biologique par lequel les cellules souches hématopoïétiques (CSH) donnent naissance à tous les éléments figurés du sang. Chez les mammifères adultes, elle se déroule dans la moelle osseuse et nécessite des interactions entre cellules hématopoïétiques et stromales au sein d’unités micro-anatomiques appelées « niches ». Durant les deux dernières décennies, le concept de « niches » a été étendu aux différents stades de différenciation des cellules hématopoïétiques suggérant que des « niches » spécifiques existent. Par ailleurs, il est admis que les cellules stromales médullaires jouent un rôle essentiel dans le vieillissement du système hématopoïétique et l’émergence d’hémopathies myéloïdes dont l’incidence augmente avec l’âge.
La thématique de recherche de l’équipe consiste à caractériser les mécanismes moléculaires impliqués dans les interactions entre cellules hématopoïétiques et stromales au sein des niches médullaires. Nos deux grands axes de recherche consistent en :
- Identification de biomarqueurs prédictifs de l’émergence d’hémopathies myéloïdes et de leur évolution en leucémies aigües myéloïdes (LAM).
- Modélisation des interactions entre cellules hématopoïétiques et cellules du microenvironnement médullaire.
Les projets reposent sur les expertises des chercheurs de l’équipe en immuno-hématologie, en biologie de l’adhérence cellulaire et en biologie clinique des syndromes myélodysplasiques (SMD) et myéloprolifératifs (SMP) qui peuvent évoluer en LAM dans 10 à 30% des cas.
Les projets et expertises
L’étude de biomarqueurs prédictifs de l’émergence et de l’évolution des hémopathies myéloïdes en LAM repose sur des analyses histologiques, phénotypiques et moléculaires d’échantillons de patients atteints de SMP, SMD ou LAM. Nous nous intéressons essentiellement à la caractérisation des cellules à l’origine des différentes pathologies, dont nous savons qu’elles accumulent un certain nombre d’altérations géniques avant de devenir pathologiques.
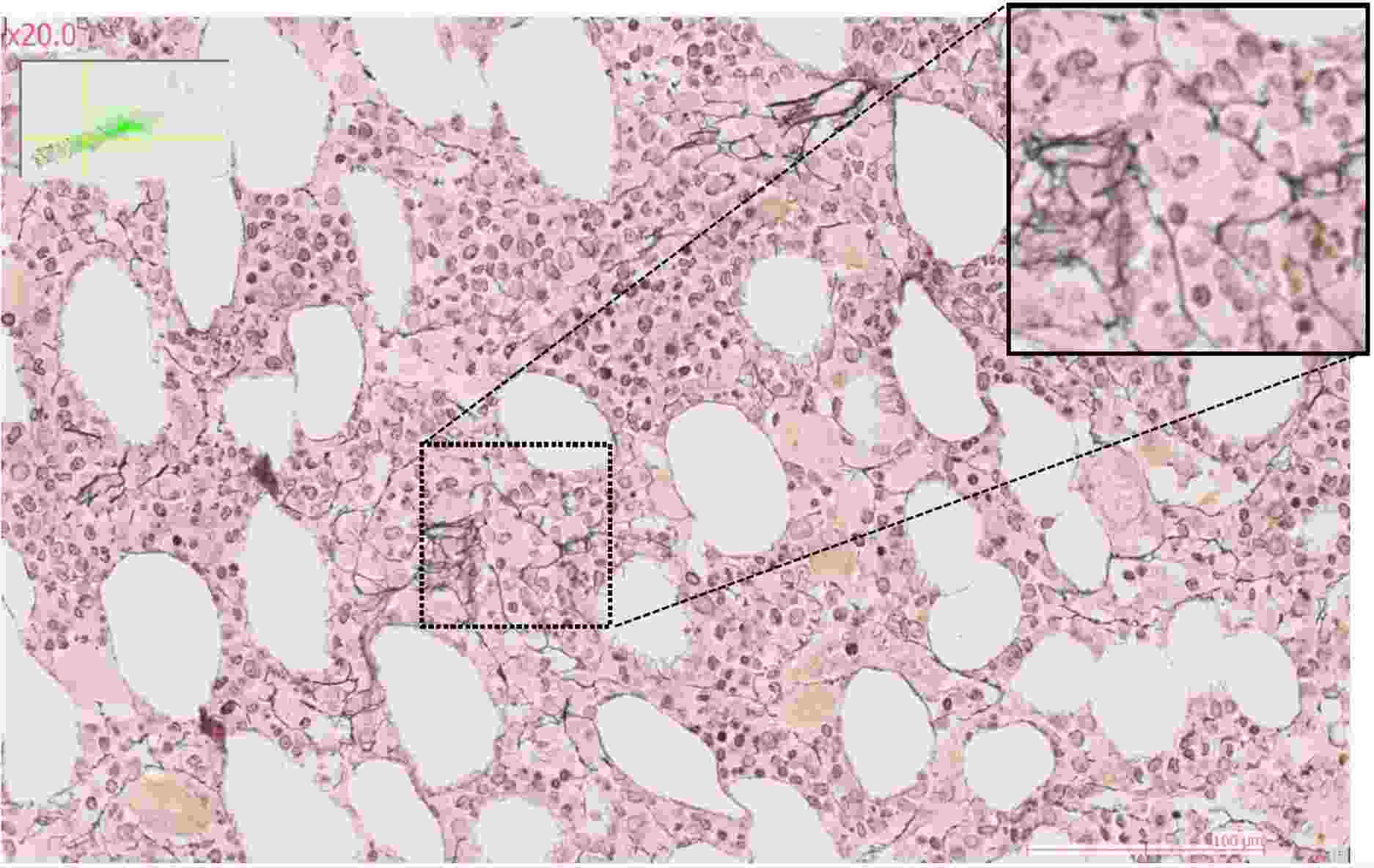
Ces altérations sont reflétées par des changements de programmes d’expression génique au niveau des cellules souches à l’apex de la pathologie, dont la conséquence est une perte de contrôle de l’homéostasie hématopoïétique. Cette perte de contrôle passe par des changements de l’organisation spatiale de la moelle osseuse chez les patients atteints de SMP ou SMD, dont certains évoluent en LAM.
La caractérisation des cellules souches malades et de leur microenvironnement est au centre de cet axe de recherche, en nous appuyant sur des données générées au sein du laboratoire que nous validons (dans la mesure du possible) sur des données publiques.
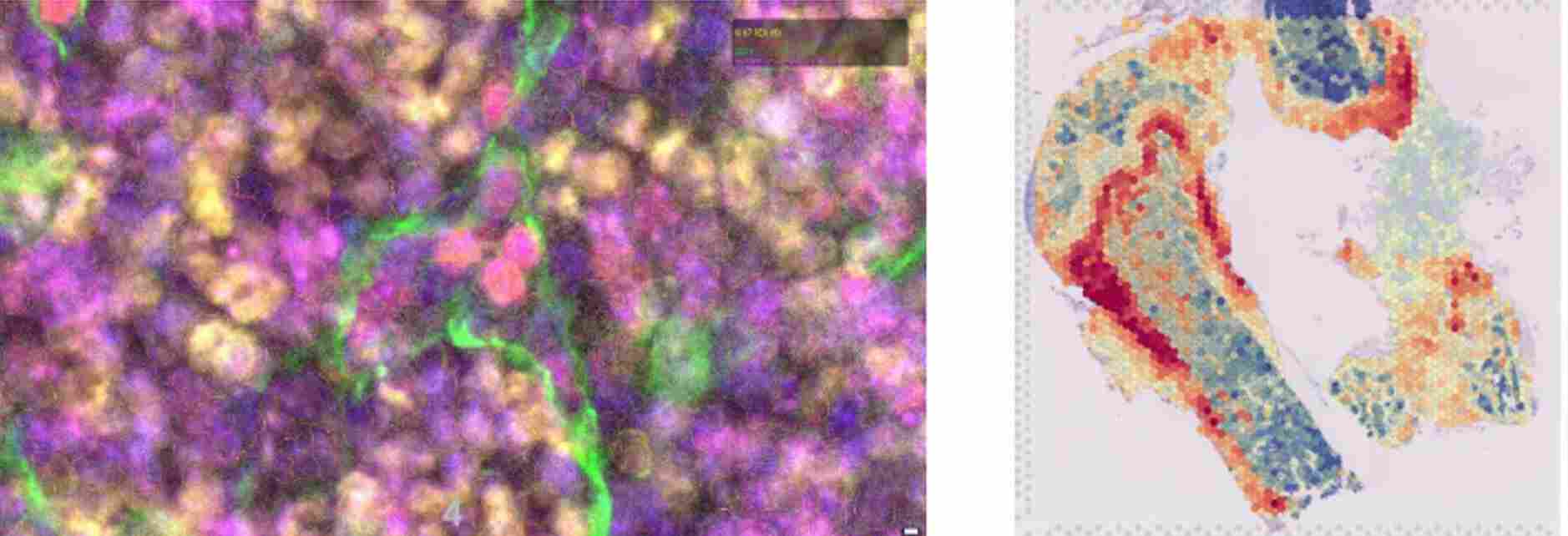
L’utilisation de technologies d’imagerie sans marquage, telles que l’imagerie de phase quantitative ou la mesure d’anisotropie (Phasics), nous permet de générer des données quantitatives d’un genre nouveau afin d’évaluer le contenu en os trabéculaire et en fibre de collagène du microenvironnement médullaire.
L’objectif ultime de cet axe de recherche est de caractériser et de visualiser in situ les réseaux d’interaction communs aux cellules d’origine des SMP et SMD qui évoluent en LAM par rapport aux cellules d’origine ne conduisant pas à cette évolution.
Afin d’étudier les modifications du stroma en condition pathologique, l’équipe s’appuie sur des modèles de leucémies aigües myéloïdes chez la souris en se focalisant sur les étapes initiales de la leucémogénèse (envahissement de la moelle osseuse inférieure à 20%). Ces modèles murins permettent de suivre la progression leucémique grâce à différentes techniques telles que la cytométrie en flux spectrale, ou d’imagerie comme le RNA-scope ou la microscopie confocale.
Afin de comprendre les modifications qui apparaissent au sein du stroma dès les débuts de la progression leucémique, nous nous basons sur des approches de transcriptomique spatiale accompagnées de cytométrie en flux spectrale (panels >25 paramètres) ou d’analyses de séquençage d’ARN en cellule unique (scRNAseq).
À ces fins, nous avons développé dans l’équipe une expertise en transcriptomique spatiale en bulk (Visium) ou au niveau de la cellule unique (Vizgen et bientôt Xenium), ainsi que les compétences bio-informatiques requises. Cet axe a pour objectif d’identifier les modifications du microenvironnement médullaire et de déterminer quelles modifications pourront être considérées comme biomarqueurs ou bien comme mécanismes de résistance aux traitements.
Parallèlement, nous nous efforçons de modéliser le microenvironnement médullaire par le biais de la fabrication de structures osseuses à base de cellules stromales, endothéliales et hématopoïétiques qui pourront être utilisées in vitro ou bien in vivo pour améliorer les modèles PDX existants qui sont peu efficaces pour greffer les hémopathies myéloïdes peu agressives ou peu proliférantes (SMD, SMP).
- Transcriptomique spatiale
- Protéomique spatiale
- Séquençage d’ARN en cellule unique
- Séquençage d’ADN en cellule unique
- Modélisation in vivo des interactions leucostromales : Ossicules
- Visualisation de coupes de tissus sans marquage (Collaboration Institut Fresnel et société Phasics)
Les actualités de l’équipe
les publications à la une
09/2024
Grenier JMP, Testut C, Bal M, Bardin F, De Grandis M, Gelsi-Boyer V, Vernerey J, Delahaye M, Granjeaud S, Zemmour C, Spinella JF, Chavakis T, Mancini SJC, Boher JM, Hébert J, Sauvageau G, Vey N, Schwaller J, Hospital MA, Fauriat C, Aurrand-Lions M.
12/2021
Bou-Tayeh B, Laletin V, Salem N, Just-Landi S, Fares J, Leblanc R, Balzano M, Kerdiles YM, Bidaut G, Hérault O, Olive D, Aurrand-Lions M, Walzer T, Nunès JA, Fauriat C.
03/2019
Balzano M, De Grandis M, Vu Manh TP, Chasson L, Bardin F, Farina A, Sergé A, Bidaut G, Charbord P, Hérault L, Bailly AL, Cartier-Michaud A, Boned A, Dalod M, Duprez E, Genever P, Coles M, Bajenoff M, Xerri L, Aurrand-Lions M, Schiff C, Mancini SJC.
09/2018
Aknoun, S., Aurrand-Lions, M., Wattellier, B., and Monneret, S.
12/2017
De Grandis M, Bardin F, Fauriat C, Zemmour C, El-Kaoutari A, Sergé A, Granjeaud S, Pouyet L, Montersino C, Chretien AS, Mozziconacci MJ, Castellano R, Bidaut G, Boher JM, Collette Y, Mancini SJC, Vey N, Aurrand-Lions M.
06/2015
Cartier-Michaud A, Bailly AL, Betzi S, Shi X, Lissitzky JC, Zarubica A, Sergé A, Roche P, Lugari A, Hamon V, Bardin F, Derviaux C, Lembo F, Audebert S, Marchetto S, Durand B, Borg JP, Shi N, Morelli X, Aurrand-Lions M.


Labels, Financements et Partenaires
Comme d'autres, ils ont fait partie de l'équipe. Merci à tous ceux qui ont contribué à l'excellence et à l'impact du CRCM.